癌症中基因组的重排对染色质折叠的影响
Abstract
基因组的构象在细胞核内是分层次的,其中TAD被认为是最能代表功能的区域,因为在TAD内部包含着可以去调控基因的调控元件,TAD边界区域的扰动可能会使相邻的TAD发生异常的交互从而去影响相邻基因的调控,相关的一些研究表明,基因组的重排现象会使得TAD的结构发生改变从而影响基因的调控,这篇文章就去探究在不同的肿瘤组织中不同的结构变异对于TAD以及基因表达的影响,从而帮助我们更好的理解染色质的折叠和基因组的重排之间的关系。
Results
TAD boundaries are affected by different types of somatic SV in cancer genomes
- 文章用了五种细胞的Hi-C数据去鉴定TAD,这五种细胞分别来自三个胚胎层:中胚层( mesoderm)、下胚层(endoderm)、外胚层(ectoderm)
- 在这五种细胞中鉴定到了3,926 to 4,690个TAD,与之前的研究数据相比有84%以上的相似度
- 之后,作者鉴定了已知的这些 TAD边界附近的特征,然后在所有细胞种一共鉴定到了2477个边界,然后这2477区域就被用于之后的分析
- 之后作者鉴定了这些边界在癌症细胞中的存在分布情况,发现在白血病细胞和乳腺癌细胞中分别有85%和83.4%的重叠,说明绝大部分边界在正常细胞和恶性的细胞中使保守的
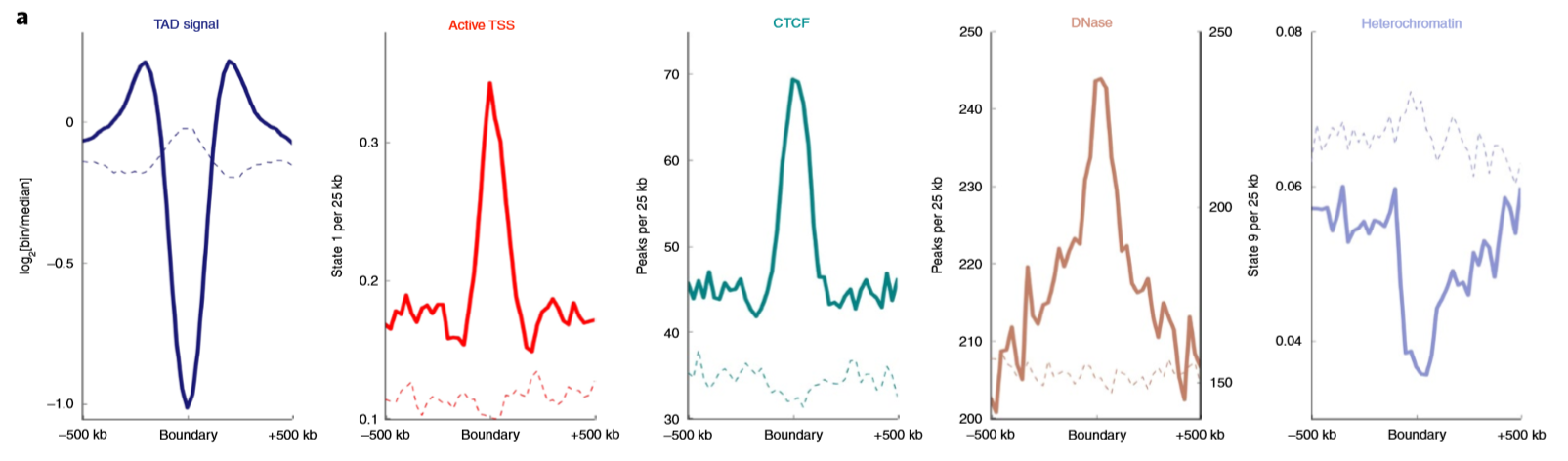
- 此外,作者发现边界TAD信号水平是最低的,这和TAD边界减少临近区域的交互这个特点相符合,如下图所示
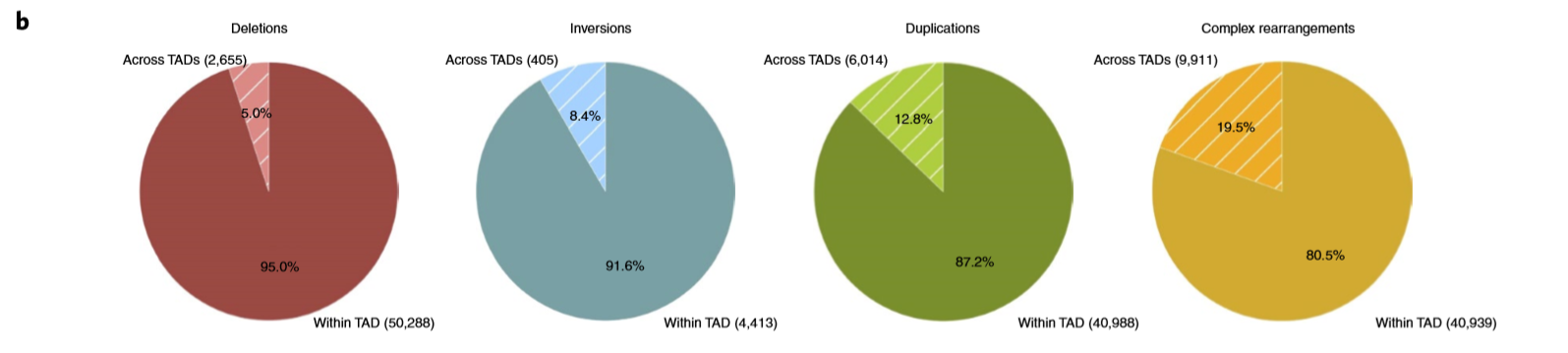
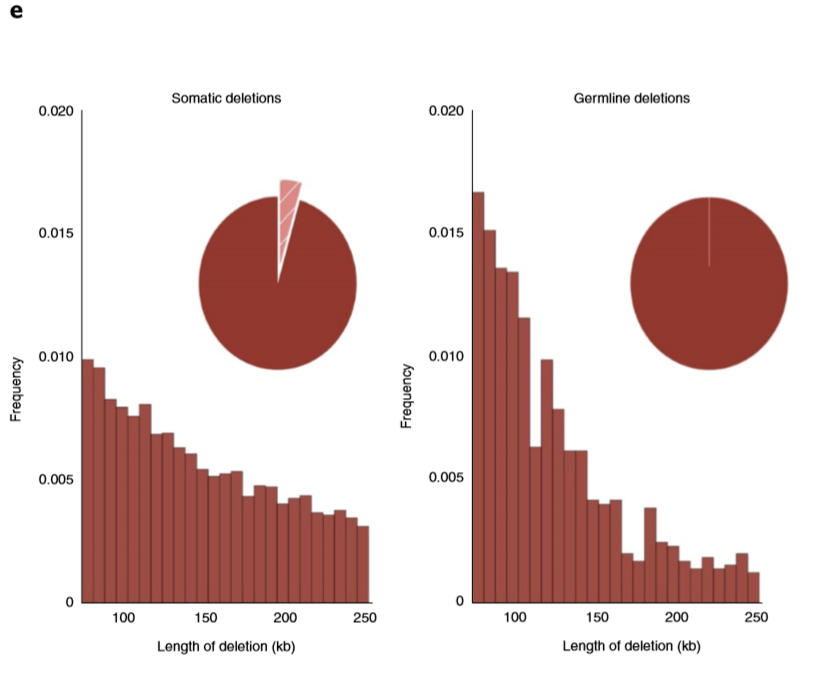
- 作者使用了288,457 个可信度较高的SVs,并且使用SVs断点的方向去将这些SVs分类(deletions, inversions, duplications or complex rearrangements),然后按照2Mb长度作为界限去将SVs分成两类:long-range SVs short-range SVs,deletions, inversions and duplications大部分都是短距离,其中大部分complex rearrangements都是长距离的,本文主要分析短距离的SVs
- 作者鉴定了那些跨越了整个TAD边界的SV,结果发现5.0%, 8.5%, 12.8% and 19.9% of all deletions, inversions, duplications and complex events是BA((boundary affecting )事件,如下图所示
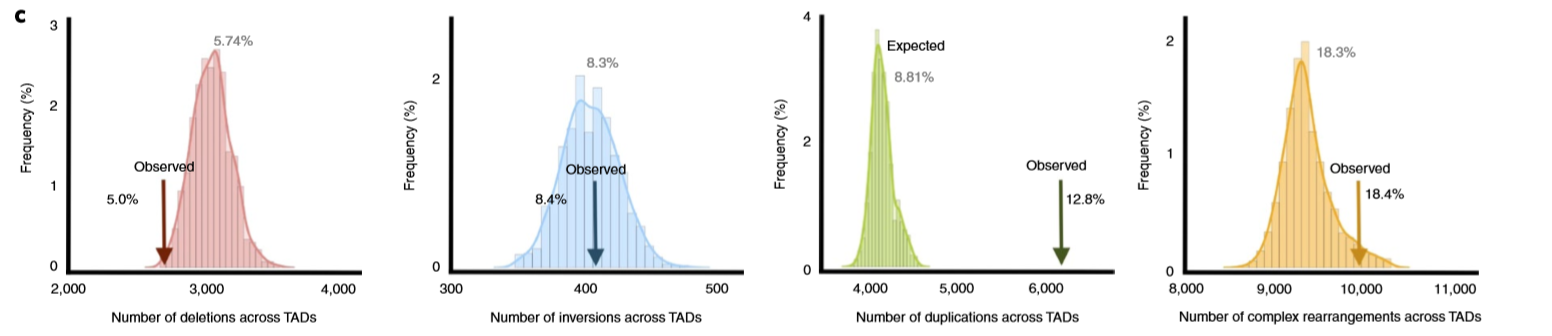
- 进而,作者发现和预期的边界扰动相比,BA-duplications高于预期,BA-deletion低于预期,其他两种在预期之内,这表明deletion更易于出现在TAD内部 ,duplication更易于跨越TAD,结果如下图
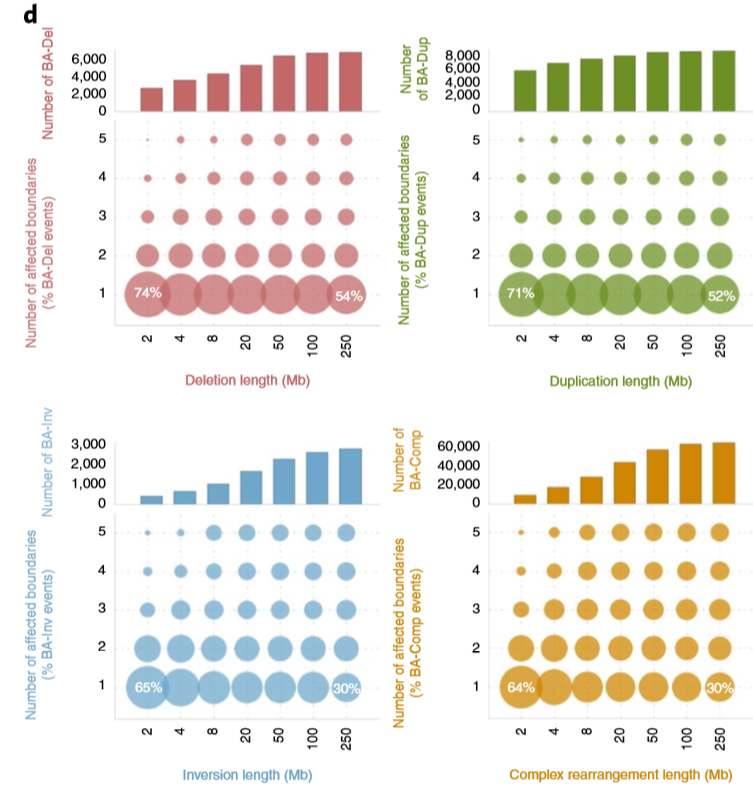
- 作者还发现,在不同的SVs种,BA-SVs在长度上的分布基本相同,大部分BA-SVs都只影响一个边界,绝大多数(98.4%) 癌细胞的TAD边界都会被影响,很有趣的是,这些TAD的边界不太可能会被健康人群基因组中的deletion and duplication所影响。图示如下
- 最后作者研究了deletions在胚胎细胞和癌细胞中的分布,发现在胚胎细胞中只有少于0.1%的deletions发生在边界上,而癌细胞中有4.1%
Chromatin folding disruptions are specific to histological subtypes
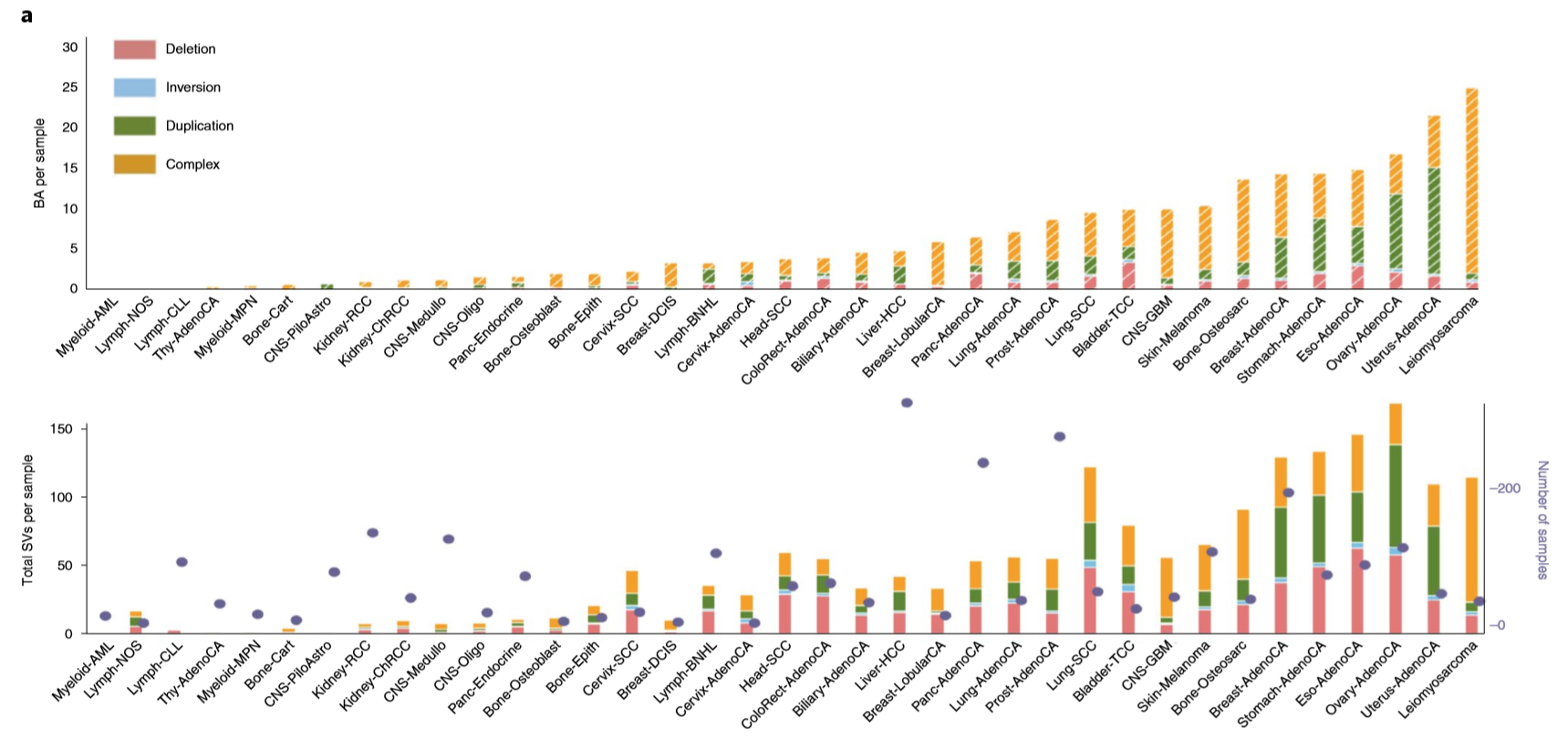
- 之后,作者比较了这些BA-SVs在不同的肿瘤类型中的分布情况,发现BA-SVs在不同的肿瘤类型之间存在显著差异
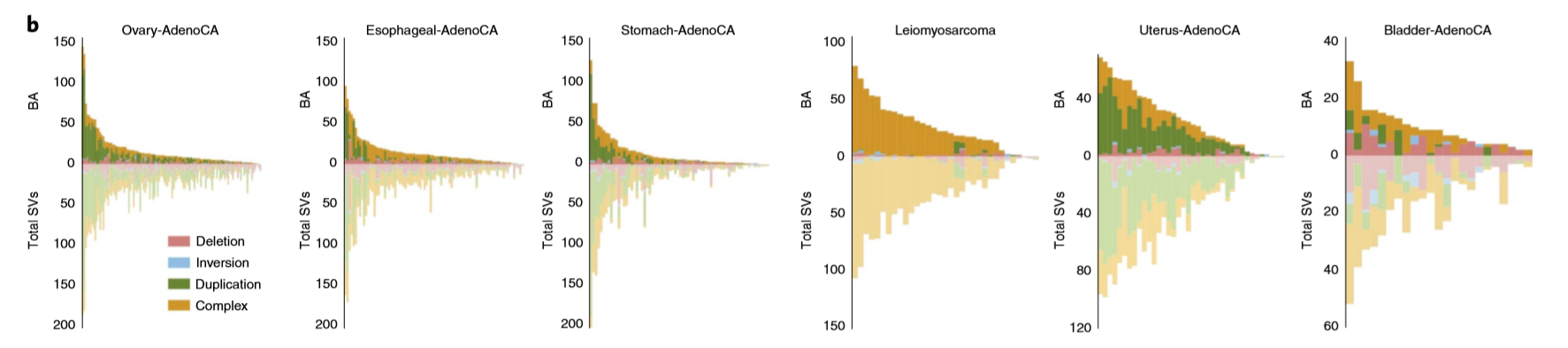
- 下面这张图描述了同一肿瘤类型的每一位病人的BA-SV分布情况
Recurrently affected boundaries in specific cancer types
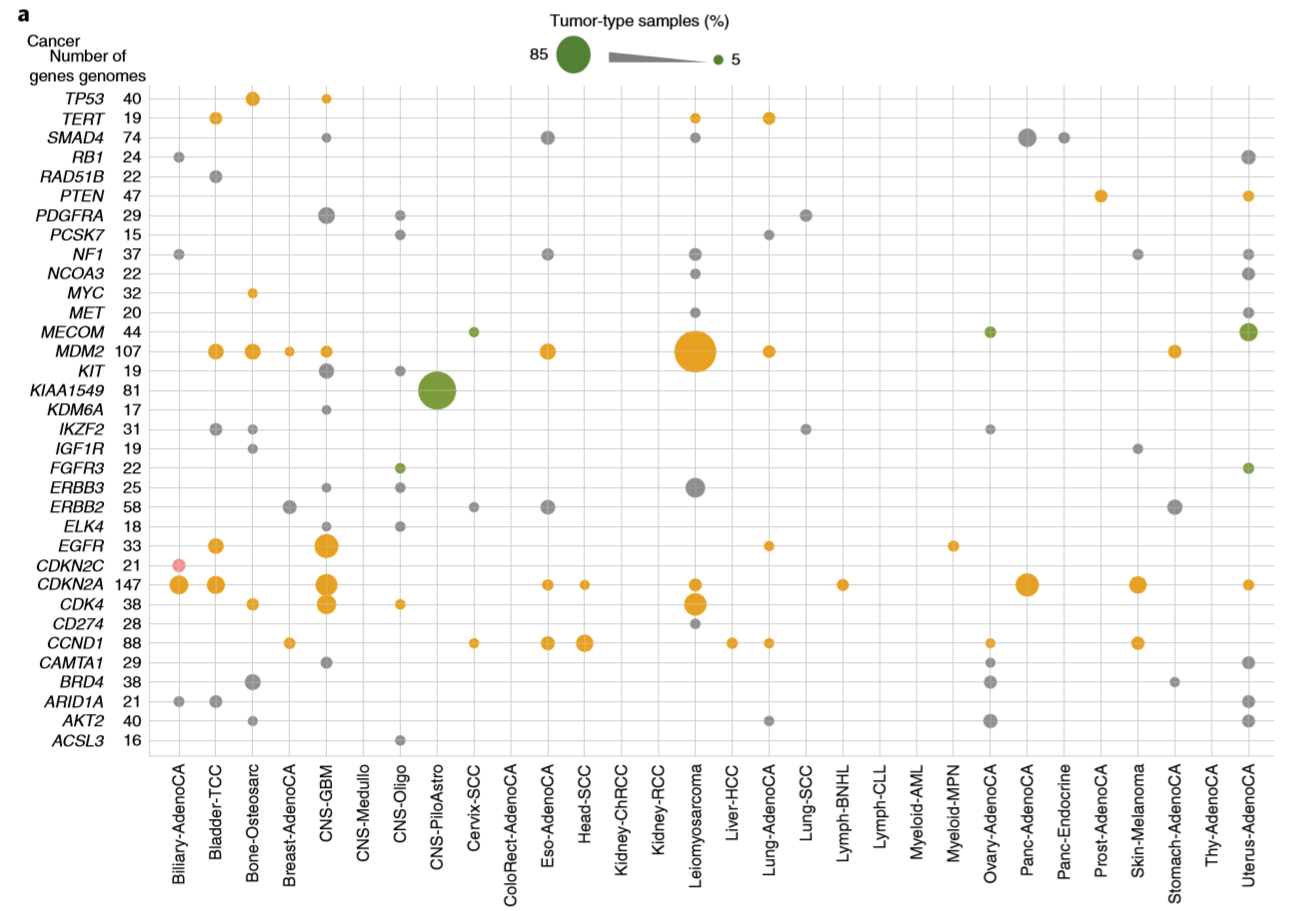
- 作者接下来探究癌症驱动基因附近的TAD边界与这些结构变异之间分布关系,结果如下(red, deletion; orange, complex; green, duplication; gray, different SV types were observed),圆圈大小代表某一个特定的肿瘤内BA-SV在这个癌症驱动基因中附近的出现比例
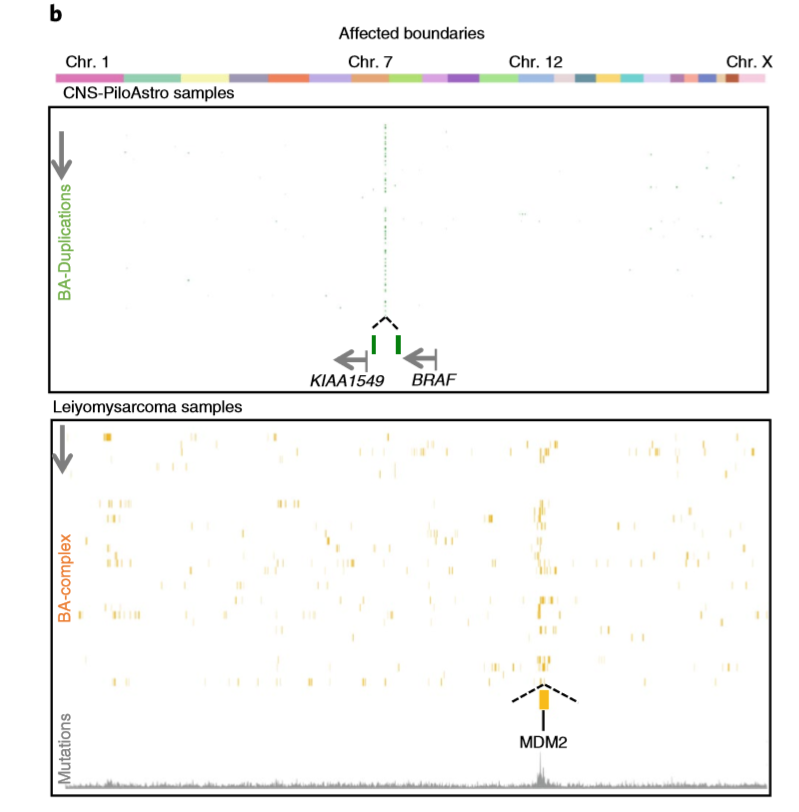
- 此外,作者发现了KIAA1549、BRAF这两个基因之间的边界在纤维性星形细胞瘤中易于发生 BA-duplications ;还发现在平滑肌肉瘤中MDM2这个位点附近的边界被影响的频率较高;同时平滑肌肉瘤12号染色体上突变频率较高,图示如下(列代表TAD边界,行代表不同的样本)
- 此外,作者研究了TAD内部的一些SVs,这些SVs主要扰动 CTCF–CTCF chromatin loops,作者鉴定了许多可能被扰动的 chromatin loops ,这表明染色质折叠的扰动会发生在不同的尺度上,包括TAD和CTCF–CTCF chromatin loop
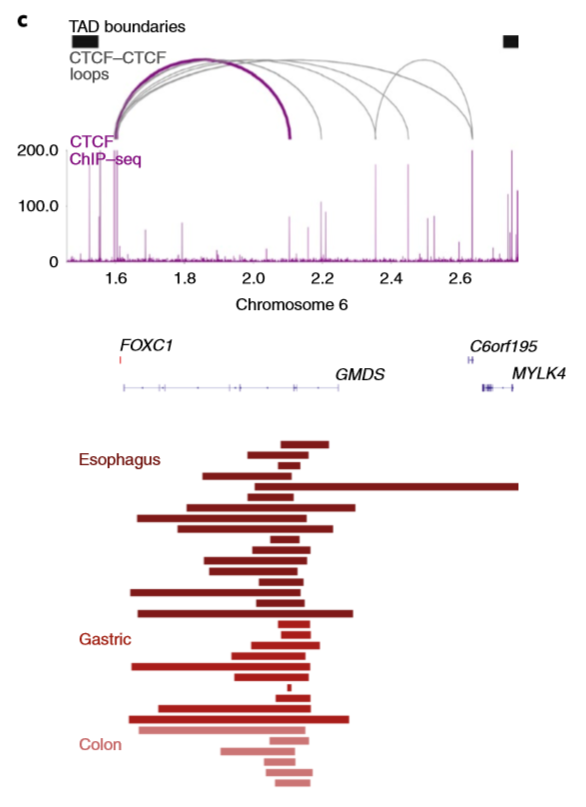
- 其中值得注意的是在食道癌、胃癌和结肠癌细胞的6号染色体上 FOXC1 基因附近的一个CTCF位点与一个删除区域有重叠,如下图所示(图中紫色的弧线新出现的CTCF–CTCF loops )
Most domain disruptions do not result in marked gene-expression changes.
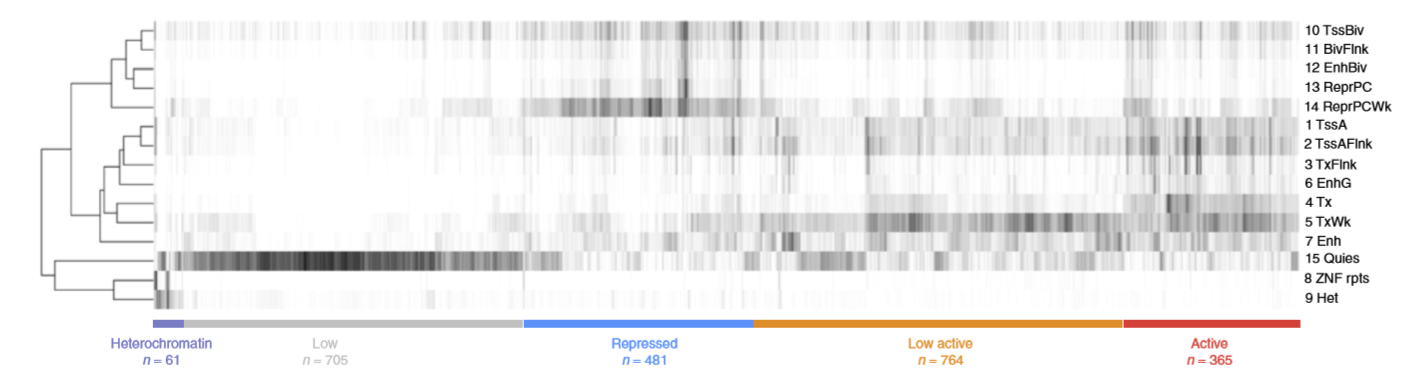
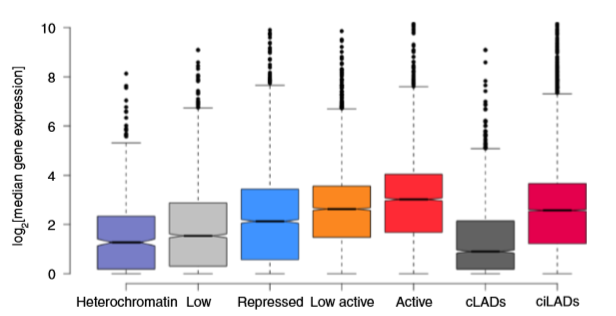
- 作者对TAD进行注释,通过分析TAD所在区域的的染色质状态将TAD分成五类: heterochromatin (61), low/quiescent (705), repressed (481), low-active (764) and active (365),然后作者计算了每种类型的TAD内部基因的表达量,发现基因在repressed domains和constitutive LADs的表达要低于active domains和inter-LADs ,图解如下
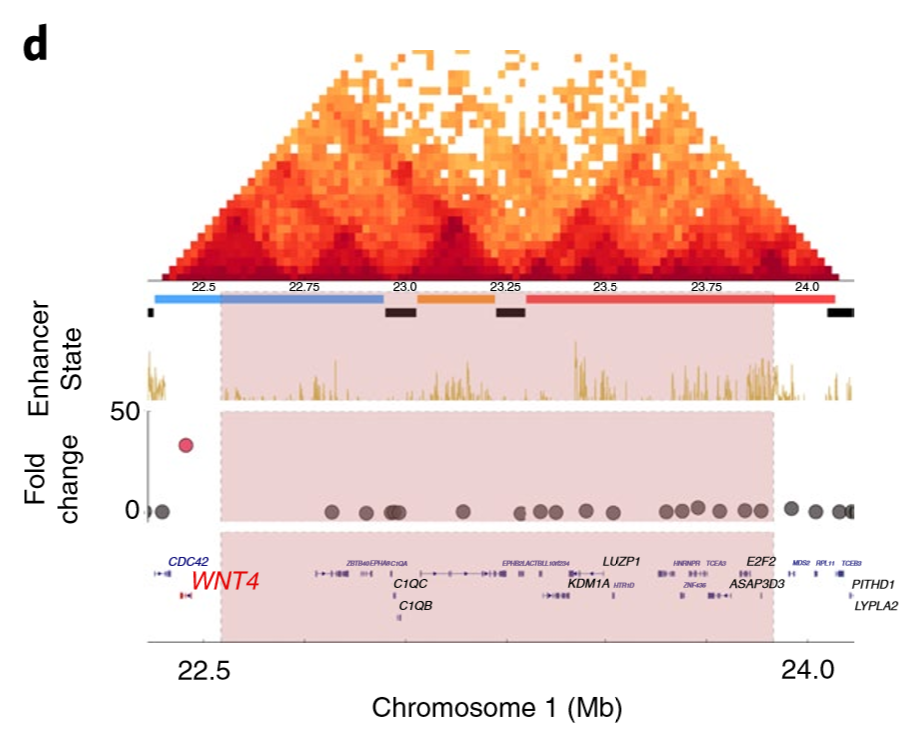
- 之后,作者研究了BA-SVs两侧的TAD的类型,发现大多数都是相同的,但不同的出现概率要大于预期,所以作者就研究了发生在活跃和抑制区域之间的BA-deletions,发现缺失导致的TAD的融合会使得附近基因表达上调,但这种现象并不是普遍存在的,图解如下
图中黑色的块代表边界,阴影部分代表 BA-deletions 区域,WNT4表达上调37倍
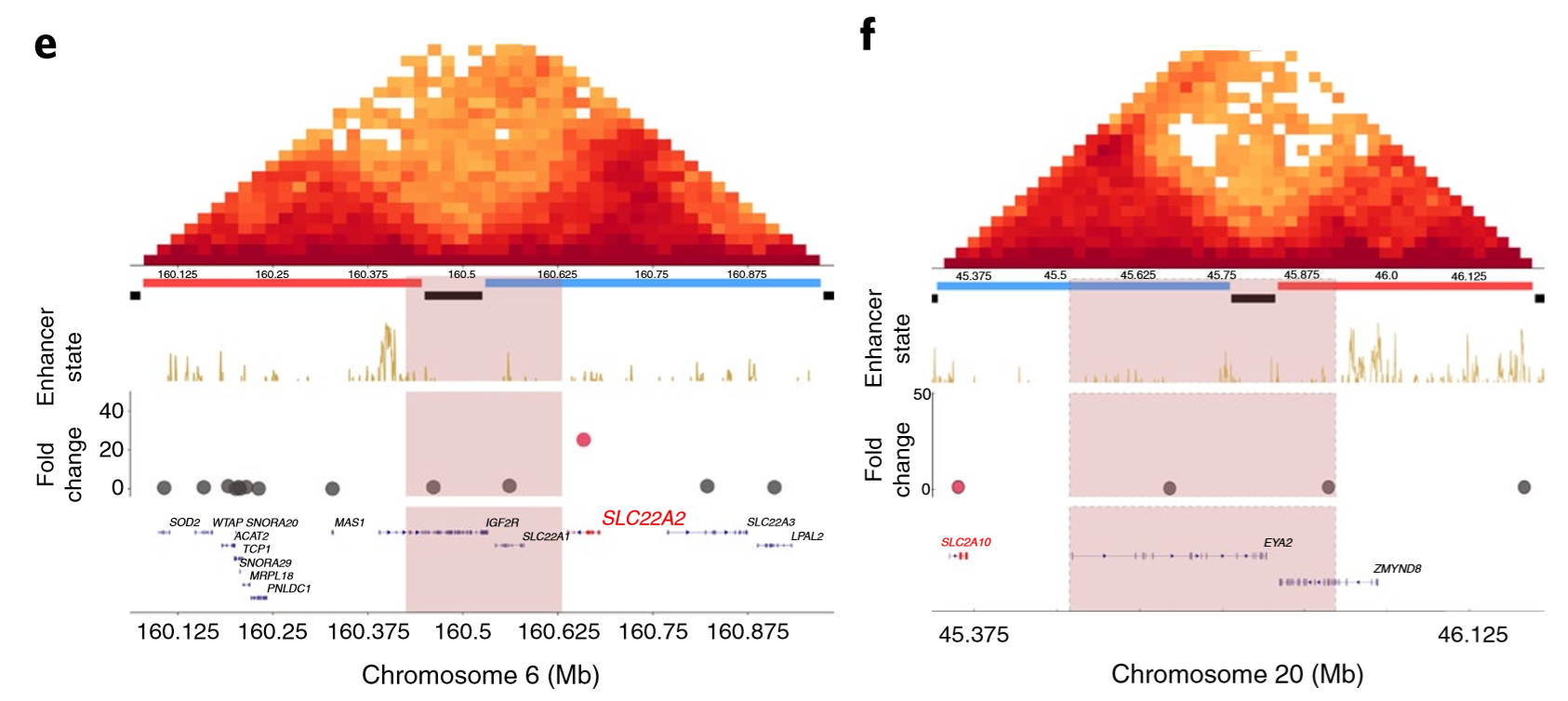
e图SLC22A2基因表达上调了26倍,但f图中SLC2A10只有1.1倍
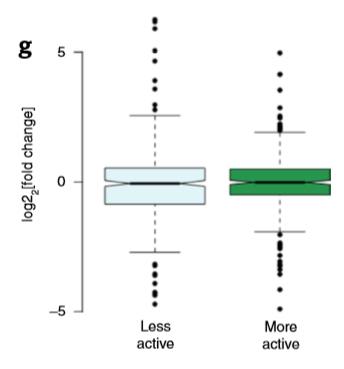
- 但作者发现整体上BA-deletions附近的基因表达并没有显著差异,只有14%发生了两倍以上的变化,图示如下
综上,作者总结出:在癌细胞基因组中基因的调控是多因素的,虽然在某些特定情况下染色质折叠的扰动会引起表达水平的差异,但并不是总是出现的。
Cell-type-specific alterations in chromatin folding patterns by different SV types.
- 作者研究了BA-deletions, BA-inversions, BA-duplications and BA-complex rearrangements 是如何影响染色质交互热图的,结果如下
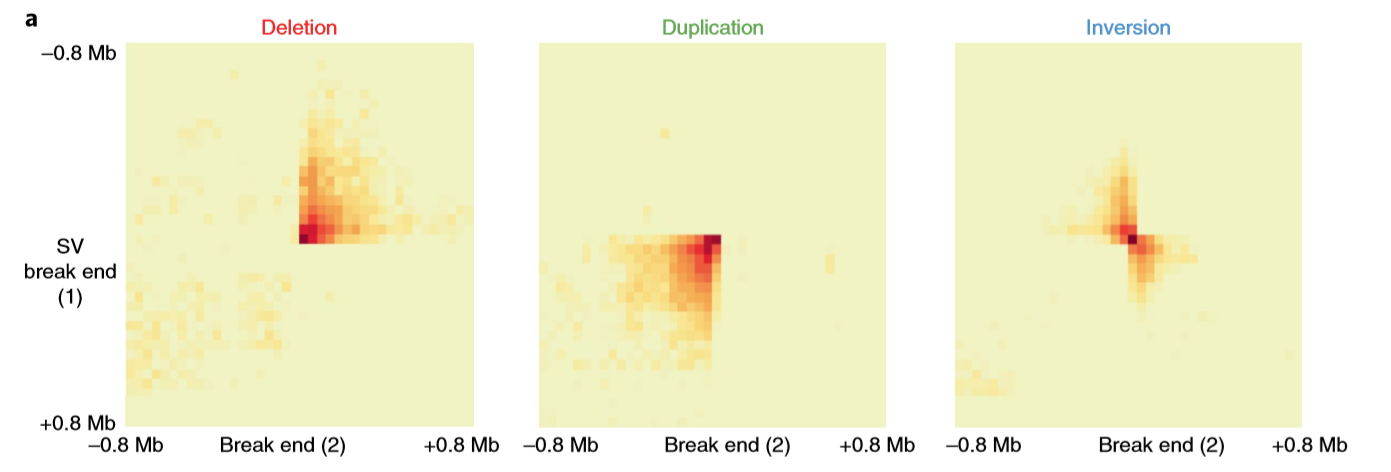
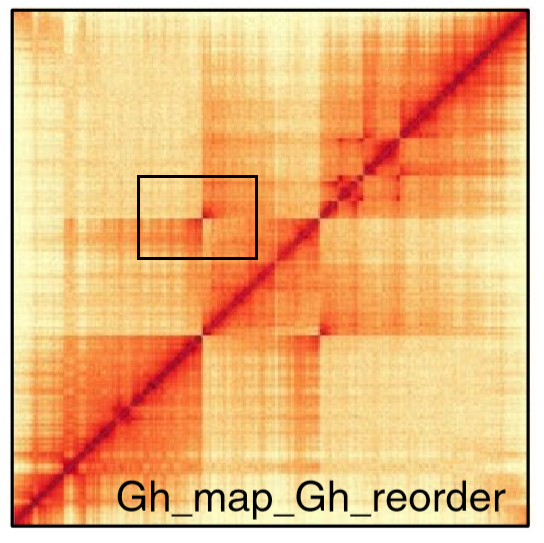
附图,图中黑色框框住的就是倒位导致的改变
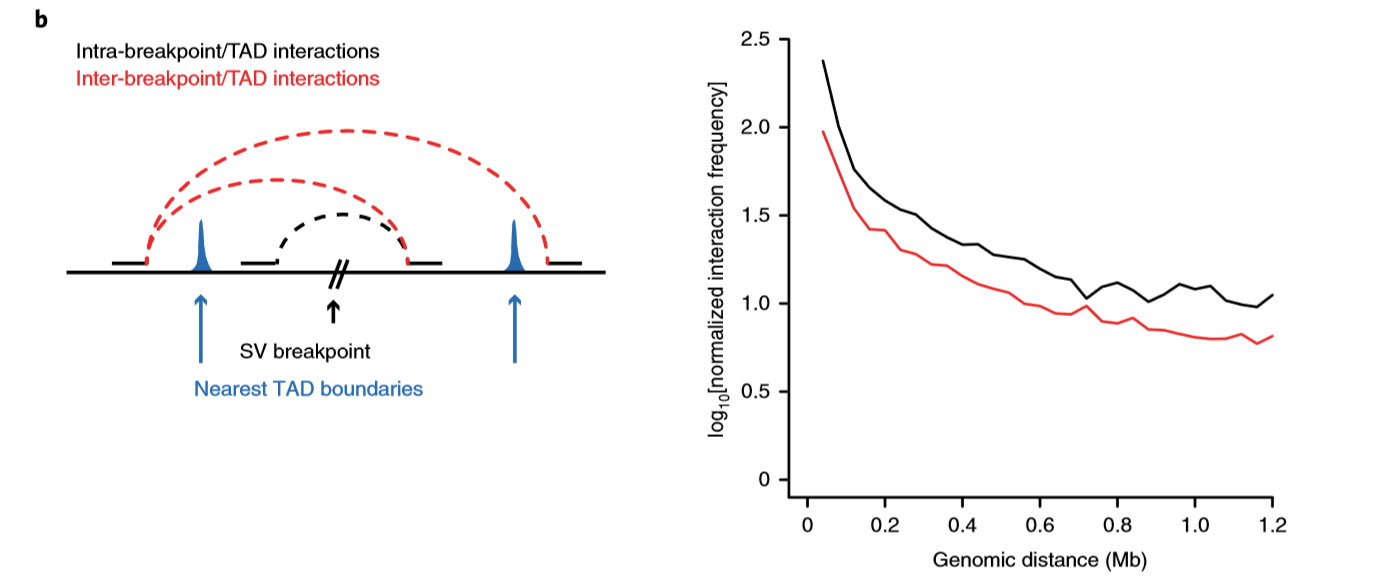
- 然后作者把跨越TAD边界和SV断点的交互称作Inter-breakpoint/TAD interactions 只跨越断点的交互称为Intra-breakpoint/TAD interactions ,作者发现后一种交互要强于前一种,所以SVs可能会导致跨越边界的交互的形成,还可能会导致新的TAD的形成。图示如下
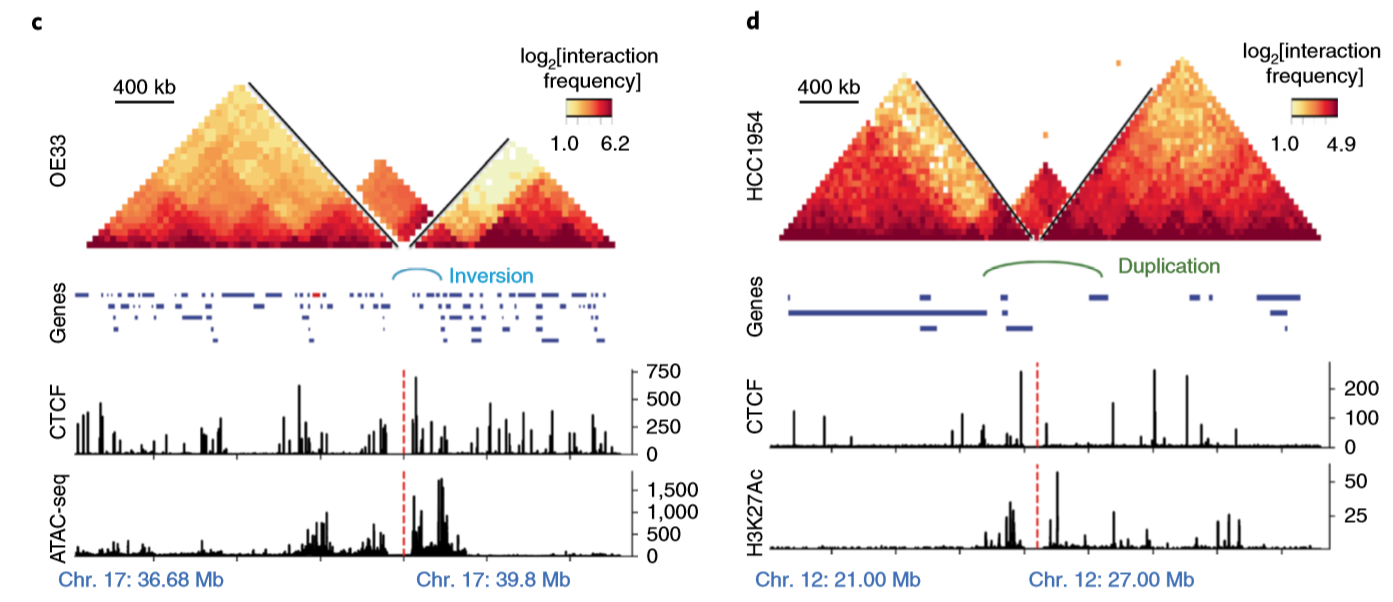
- 作者举了两个TAD改变的例子
左图是倒位导致的TAD的融合,右图是加倍导致的TAD的形成
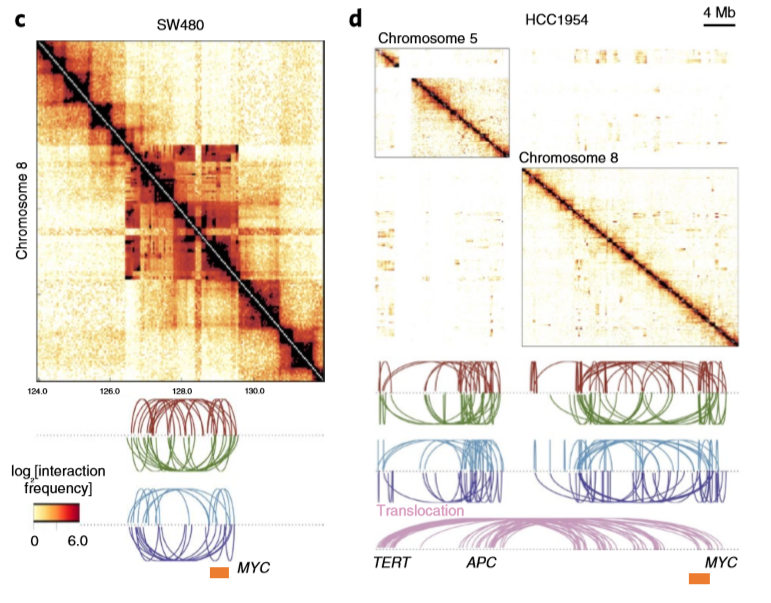
Complex rearrangements markedly change chromatin folding maps in the cancer genomes
- 复杂的重排现象会明显导致Hi-C热图发生明显的变化,如下图所示:左图展示了8号染色体上的一个复杂的重排区域( Green, tandem duplication; red, deletion; cyan and purple, inversion),右图展示了5号和8号染色体上以及两个染色体间的之间复杂的重排区域导致的热图的变化
Discussion
- 本文作者探讨了SVs在多种肿瘤类型中的分布及其在染色质折叠和基因调控中的潜在作用。
- 其次作者发现,一些边界的改变只分布在某一特定的癌症中,这可能与一些特定的癌症驱动基因有关
- 删除易于发生在TADs和LADs内,而加倍倾向于发生在TADs之间,这表明不同类型的SVs的产生机制可能存在差异
- 值得注意的是,作者没有观察到TAD的扰动与基因的总体表达水平变化之间有很强的关联,只有14%的TAD扰动导致了2倍以上的表达上调
参考
Disruption of chromatin folding domains by somatic genomic rearrangements in human cancer

