circos进阶之link图
背景
Links主要试图利用连线关系,表明染色体上不同位置之间的联系。这在实际生物学问题中很常用,尤其是在表示基因和基因之间的共线性的时候,link图是我们的不二选择,之前我们一起学习了使用MCScanX进行共线性分析,下游分析中可以画一个圈图,但是本人认为那个圈图无论是配色还是布局都不是特别好看,今天我们就来学习如何处理MCScanX的结果文件然后用circos去画出我们想要的圈图
本次教程相应的配置文件和原始数据文件,请点击此处下载
文件的处理
link图是需要输入数据的,所以下里面介绍一下link图的输入数据格式
1
2
3
4
5
| ...
hs1 100 200 hs2 250 300 color=ch1
hs2 400 550 hs3 500 750 color=ch2
hs3 600 800 hs4 150 350 color=ch3
...
|
我们来看看我们的raw data
1
2
3
4
5
| 0- 0: Ghir_A01G000050.1 Ghir_D01G000050.1 2e-52
0- 1: Ghir_A01G000080.1 Ghir_D01G000080.1 5e-171
0- 2: Ghir_A01G000090.1 Ghir_D01G000100.1 2e-26
0- 3: Ghir_A01G000100.1 Ghir_D01G000120.1 0
0- 4: Ghir_A01G000110.1 Ghir_D01G000130.1 9e-179
|
首先我们要找出MCScanX的结果文件At-Dt.collinearity,通过gff文件,把每一个基因的注释信息找出来,指令如下
1
2
3
| awk -F "\t" '{print $2 "\t" $3}' At-Dt.collinearity | awk '/^Ghir/{print $1}' | xargs -I {} grep {} ./At-Dt.gff | awk '{a=substr($2,1,8)}{print a "\t" $3 "\t" $4}' > 1.tmp
awk -F "\t" '{print $2 "\t" $3}' At-Dt.collinearity | awk '/^Ghir/{print $2}' | xargs -I {} grep {} ./At-Dt.gff | awk '{a=substr($2,1,8)}{print a "\t" $3 "\t" $4}' > 2.tmp
paste 1.tmp 2.tmp >link.tmp
|
为了区分不同染色体之间的link,我们这里让link的染色和第一条染色体颜色相同,我们引入一个参数去定义颜色,指令如下
1
| awk '{a=substr($1,7,2)}{print $0 "\t" "value=rgb"a}' link.tmp >link.tmp1
|
我们的link文件就变成了
1
2
3
4
5
| Ghir_A01 76870 77973 Ghir_D01 33573 38297 value=rgb01
Ghir_A01 88691 90135 Ghir_D01 58267 58998 value=rgb01
Ghir_A01 93974 95093 Ghir_D01 67754 69206 value=rgb01
...
Ghir_A13 46708701 46711385 Ghir_D13 21207310 21209912 value=rgb13
|
这里我们再使用linux指令对我们的link文件进行筛选,指令如下
1
| awk '{gsub(/A0/,"A",$1);gsub(/D0/,"D",$4);print $0}' OFS="\t" link.tmp|awk '{a=substr($1,7);b=substr($4,7);if(a==b){print $0}else if(a>=2&&a<=3&&b>=2&&b<=3){print $0}else if(a>=4&&a<=5&&b>=4&&b<=5){print $0}}'|awk '{gsub(/A([0-9])$/,"0&",$1);gsub(/D([0-9])$/,"0&",$4);print $0}' OFS="\t" |sed -e 's/0A/A0/g' -e 's/0D/D0/g' >link.txt
|
这里我们筛选条件有两个
配置文件的书写
circos.conf、ideogram.conf、karyotype.conf、ticks.conf之前的文章有讲述,这里只是多了一个links和colors模块,所以我们这次重点讲述这两个模块
mycolors.conf
染色体文件karyotype.txt如下
1
2
| chr - Ghir_A01 At01 0 117757855 rgb01
chr - Ghir_A02 At02 0 108092100 rgb02
|
看到这里肯定有许多小伙伴不太明白最后一列rgb的意义,我们就来看一下前面引用的colors.conf
1
2
3
4
5
| rgb01=26,188,156
rgb02=46,204,113
...
rgb12=192,57,43
rgb13=142,68,173
|
这里对每一个rgb都进行了注释,这里采用的注释信息均来自于flatuicolors,可以直接在上面复制一些自己喜欢的颜色的rgb值。
links.conf
下面我们就来看一下links.conf这个控制文件
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
| <links>
<link>
# 指定link文件,最好使用绝对路径
file=/public/home/yxlong/yxlong/synteny/At-DtMCScanX/circos/link.txt
# links的起始终止位置
radius=0.95r
color=black_a4
# Circos提供bezier_radius、crest、bezier_radius_purity等参数来控制link的弯曲形式,具体见下图
bezier_radius=0.1r
thickness=2
# record_limit = 控制最后图片上显示的link条数
record_limit =
# 一下就是rules的使用了,这里主要对于link的颜色进行指定,使得link的颜色和A亚组的染色体颜色相同
<rules>
<rule>
condition= var(value) eq "rgb01"
color=rgb(26,188,156)
</rule>
<rule>
condition= var(value) eq "rgb02"
color=rgb(46,204,113)
</rule>
<rule>
condition= var(value) eq "rgb03"
color=rgb(52,152,219)
</rule>
<rule>
condition= var(value) eq "rgb04"
color=rgb(155,89,182)
</rule>
<rule>
condition= var(value) eq "rgb05"
color=rgb(52,73,94)
</rule>
<rule>
condition= var(value) eq "rgb06"
color=rgb(241,196,15)
</rule>
<rule>
condition= var(value) eq "rgb07"
color=rgb(230,126,34)
</rule>
<rule>
condition= var(value) eq "rgb08"
color=rgb(231,76,60)
</rule>
<rule>
condition= var(value) eq "rgb09"
color=rgb(149,165,166)
</rule>
<rule>
condition= var(value) eq "rgb10"
color=rgb(243,156,18)
</rule>
<rule>
condition= var(value) eq "rgb11"
color=rgb(211,84,0)
</rule>
<rule>
condition= var(value) eq "rgb12"
color=rgb(192,57,43)
</rule>
<rule>
condition= var(value) eq "rgb13"
color=rgb(142,68,173)
</rule>
</rules>
</link>
</links>
|
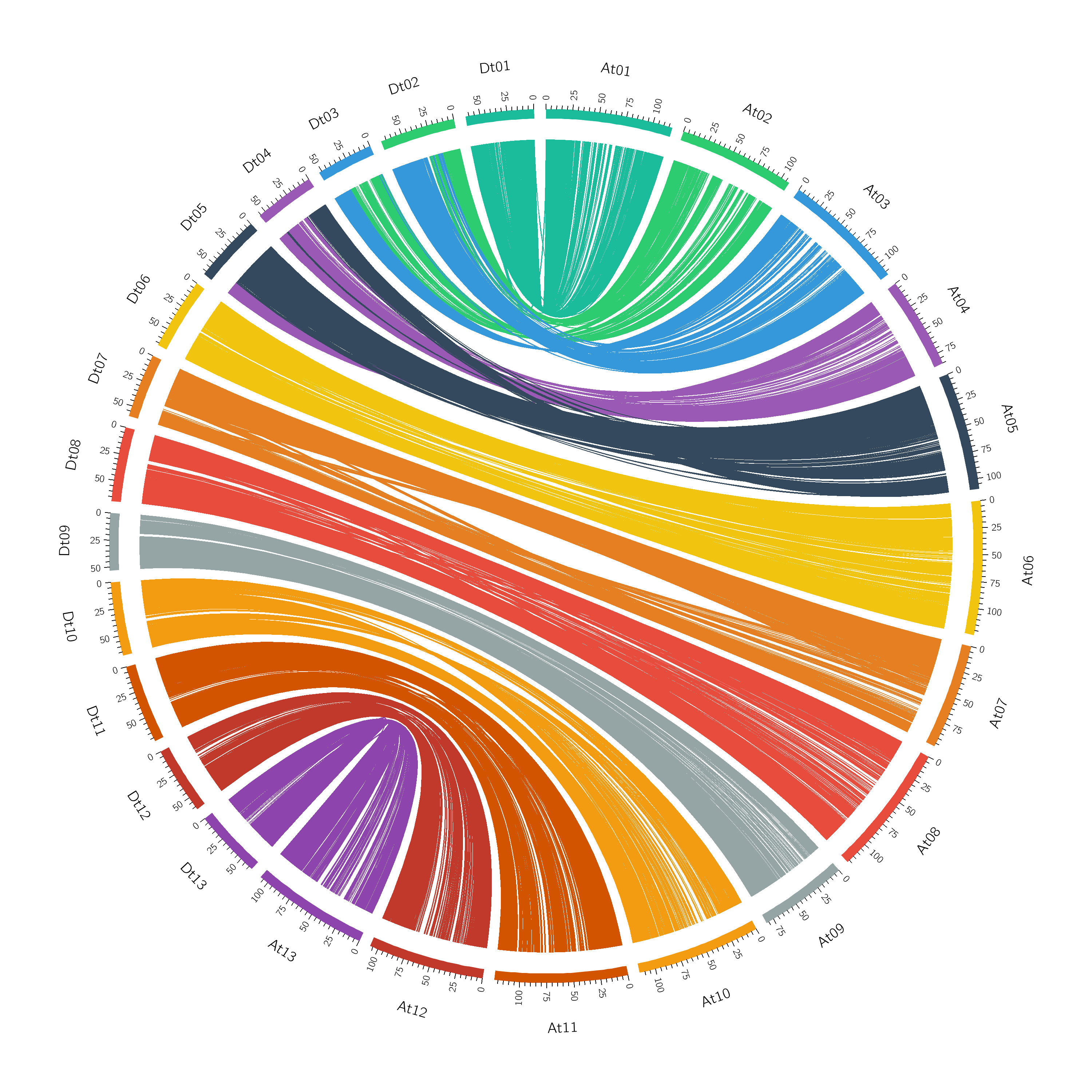
最后我们输入指令circos -conf circos..conf等待一会儿就可以得到下面这张图片
![G93ZS1.png]()
我们的circos之links图就画到这里了,之后我们继续学习更多的circos功能