circos的简介
众所周知,生物信息学是一个以庞大的数据量为基础的学科,这也就是使得结果的可视化变得有些困难,随着生物信息的发展,其辅助分析工具也迅速增多,Martin Krzywinski等人开发的Circos就在一定程度上解决了可视化困难这个问题。Circos是一个可视化数据和信息的软件包。它以圆形布局可视化数据——这使得Circos非常适合探索对象或位置之间的关系。它是创建高质量信息图表和插图的理想工具,具有丰富的分层数据和令人愉快的对称性。你可以根据你的受众调整其焦点和细节并且可以很好地控制图中的每个元素,此外,其具备高度自动化的特点,是由纯文本配置文件控制,十分的方便快捷。
circos的安装
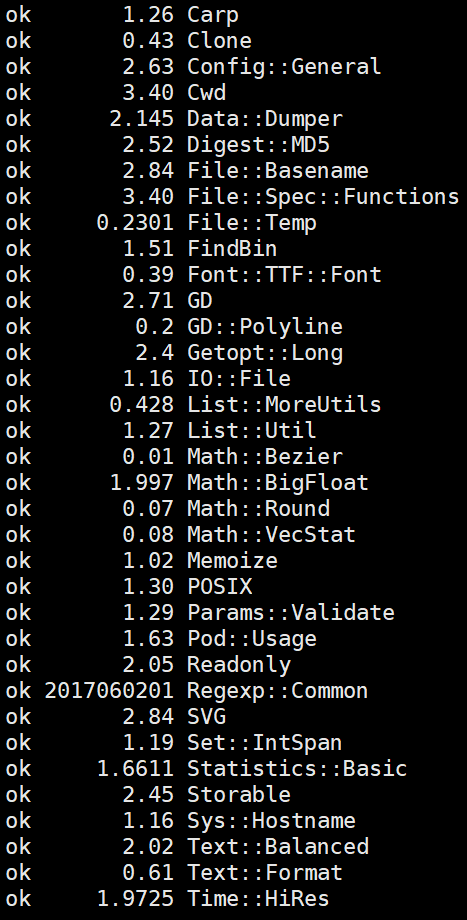
首先在这里下载到最新的Circos软件包,这里主要介绍linux版本,Circos本身是基于Perl的,所以不需要编译安装。解压下载的软件包到你想要存放的位置,并将circos/bin加入环境变量即可。它依赖较多的Perl模块,可以用circos -module查看相关Perl模块的安装情况。如果全部安装完毕,结果如图所示
![8RAyfP.png]()
如果安装模块儿出现问题,别着急,之后会有文章详细介绍模块儿的安装哦
样本的运行测试
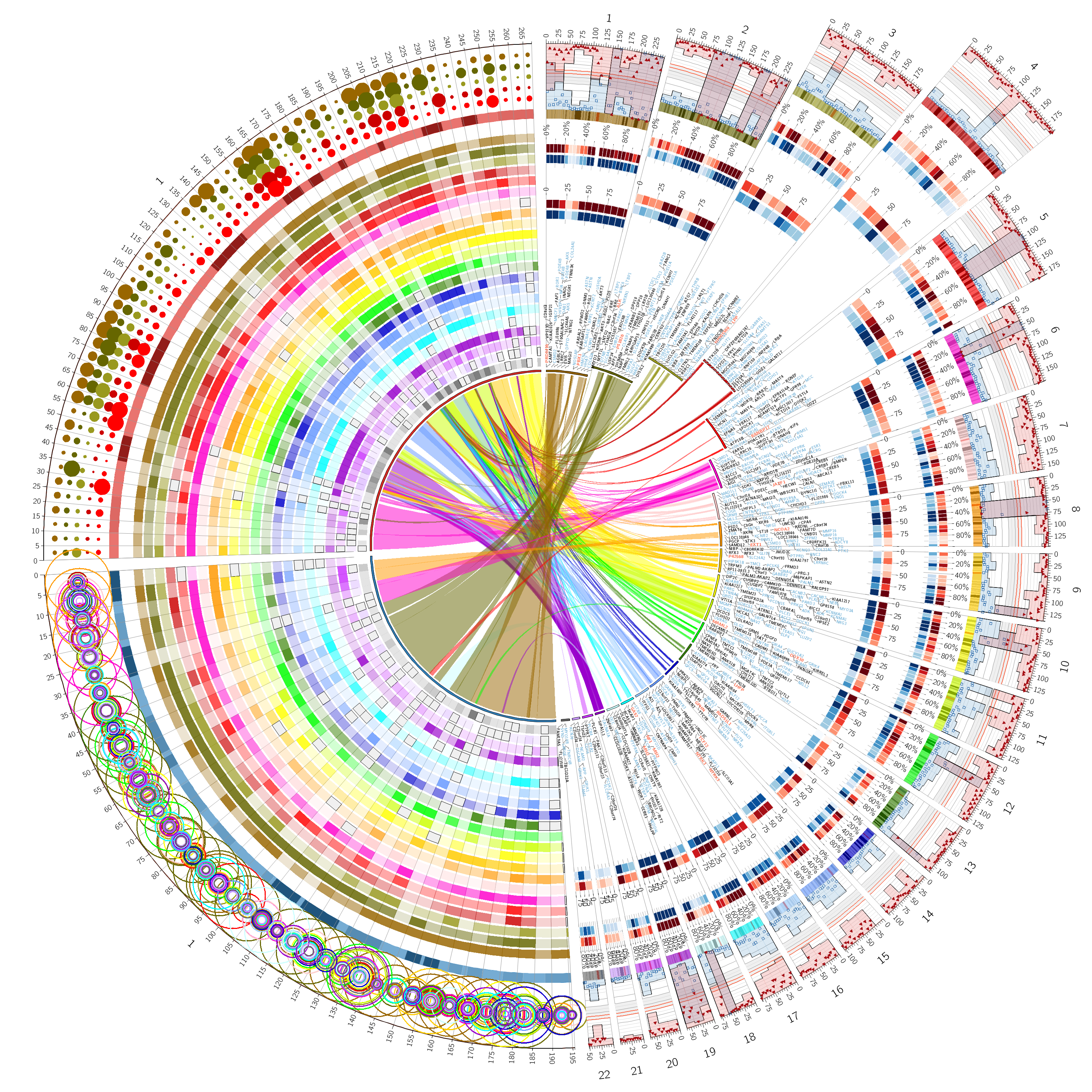
Circos包里有自己的demo,可通过circos -conf yourpath/circos/example/etc/circos.conf来运行,如果你成功的在命令行当前目录下生成png和svg两个图形文件。如下图所示,那么恭喜你已经完成了第一步并且得到了一个十分漂亮的图。
![8RAIkn.png]()
数据文件格式
circos主要有四个模块儿需要输入数据文件,包括karyotype以及highlight、plot、link,其格式都相对简单,编辑起来也不麻烦。
我们来看karyotype这个数据文件,这个文件记载了染色体的相关信息,其基本数据格式如下
1
2
3
4
5
6
7
| chr - ID LABEL START END COLOR
chr - hs1 1 0 249250621 chr1
chr - hs2 2 0 243199373 chr2
chr - hs3 3 0 198022430 chr3
chr - hs4 4 0 191154276 chr4
chr - hs5 5 0 180915260 chr5
...
|
这里第一列和第二列一般都是固定的
ID是染色体的名称,一定是你课题中使用的染色体名称
LABEL是在图中显示的染色体名称
START、END尽量覆盖全长,其他的控制文件可以控制画出来的长度
最后一列的color可以随心所欲的自己控制
配置文件的书写
最简配置文件
配置文件至少需要五个部分,
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
| # Karyotypes
karyotype = data/karyotype/karyotype.human.txt
# Ideograms
<ideogram>
<spacing>
default = 0.002r
</spacing>
thickness = 25p
fill = yes
radius = 0.80r
</ideogram>
# Image
<image>
<<include etc/image.conf>>
</image>
# RGB/HSV color definitions, color lists, location of fonts, fill patterns.
<<include etc/colors_fonts_patterns.conf>>
# Debugging, I/O and other system parameters
<<include etc/housekeeping.conf>>
|
我们可以在…/circos-0.69-9/example/etc/下面创建一个test文件夹来逐渐学习我们的配置文件,在test文件夹下创建一个新的circos.conf文件,并将上述代码复制进去,并执行下列指令
1
| circos -conf ./circos.conf
|
就可以得到这样的一张图片
![8RAcSf.png]()
下面我们就对上述代码中涉及到模块儿进行学习,包括karyotype、ideogram、ticks等模块。
Karyotype
Karyotype的信息就如同坐标轴一般,其大小、顺序、位置等直接决定了后续数据的展示。这里我们新建一个karyotype.conf文件用来设置karyotype的相关信息,主要设置的参数包括数据文件来源、是否使用特定染色体、染色体显示的单位大小(unit)、颜色、图中半径等,我们可以在我们的circos.conf中使用<>来进行调用,karyotype.conf文件的具体格式如下
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
| # 指定数据文件的位置,这里共使用了人类的数据
karyotype = data/karyotype/karyotype.human.txt
chromosomes_display_default = no
# 使用哪些染色体,这里可以使用正则表达式
chromosomes = /hs[12345678XY]$/ #匹配以[]中的字符结尾的
# 染色体排序方式
chromosomes_order_by_karyotype = yes
# 染色体单位大小,在后续进行ticks等的设置时都会参考
chromosomes_units = 1000000
# 设置染色体颜色,不指定则用默认设置
chromosomes_color =
# 将染色体反向排列,将所需反向排列的染色体名称写在下面即可
chromosomes_reverse = hs1
# 改变染色体在circos环图内所占比例
chromosomes_scale = hs1:0.25r;hs2:0.25r
# 改变染色体在circos环图内半径大小,如图中右上角
chromosomes_radius = hs1:1.15r;hs2:1.05r
|
图片会变成下面这个样子
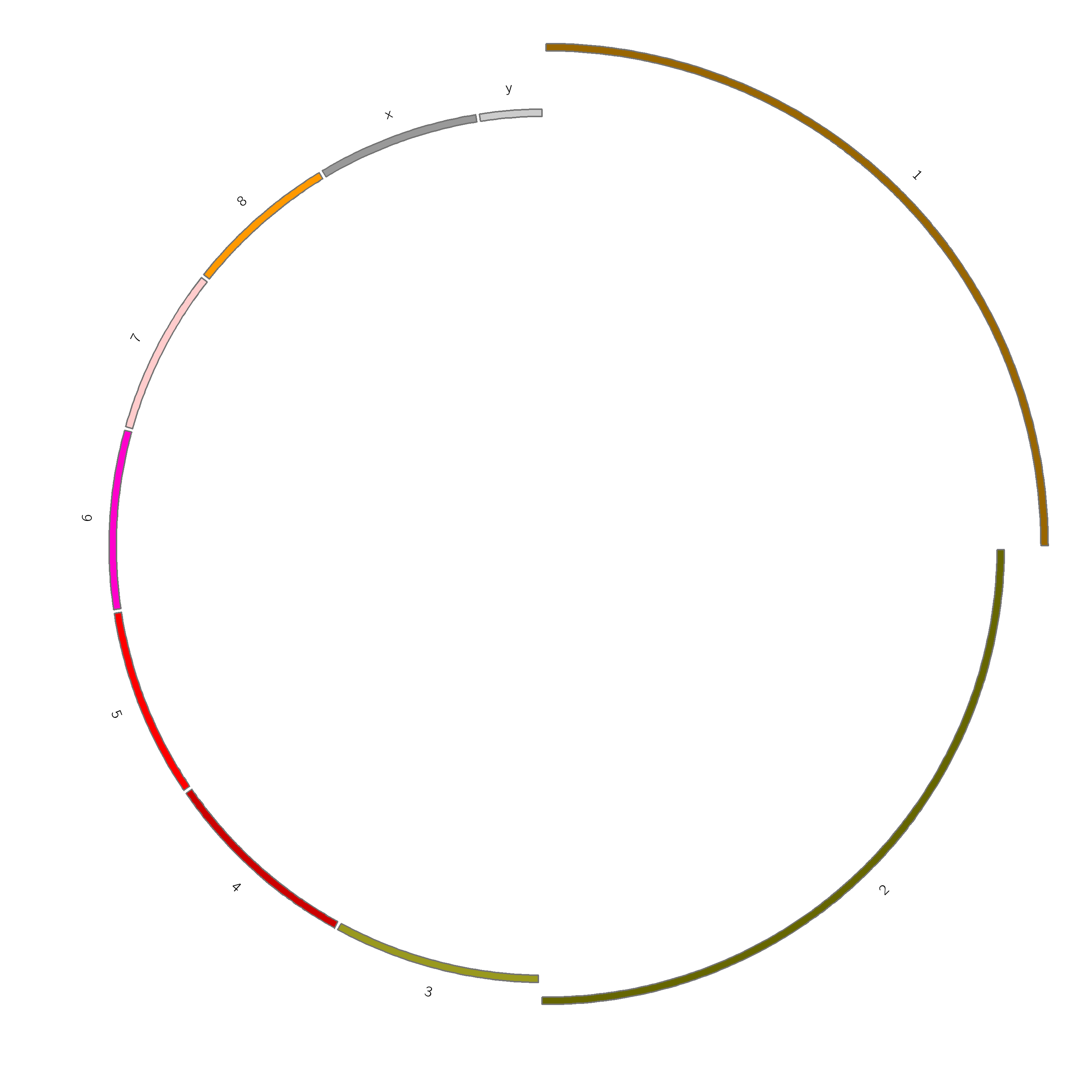
![8RAhwj.png]()
Ideogram
Ideogram主要是karyotype相关的其它一些基本显示信息的设置,包括线条颜色、粗细、间隔,标签位置、字体等,这里我们新建一个ideogram.conf文件用来管理ideogram的相关信息。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
| <ideogram>
<spacing>
default = 0.002r
</spacing>
# Ideogram position, fill and outline
radius = 0.80r
thickness = 20p
fill = yes
stroke_color = dgrey
stroke_thickness = 4p
#显示label
show_label = yes
label_font = default
## label位置在圈外侧50像素
label_radius = dims(ideogram,radius_outer) + 50p
label_size = 30
# label与karyotype平行
label_parallel = yes
</ideogram>
|
我们的圈图就变成了下面这个样子
![8RAROg.png]()
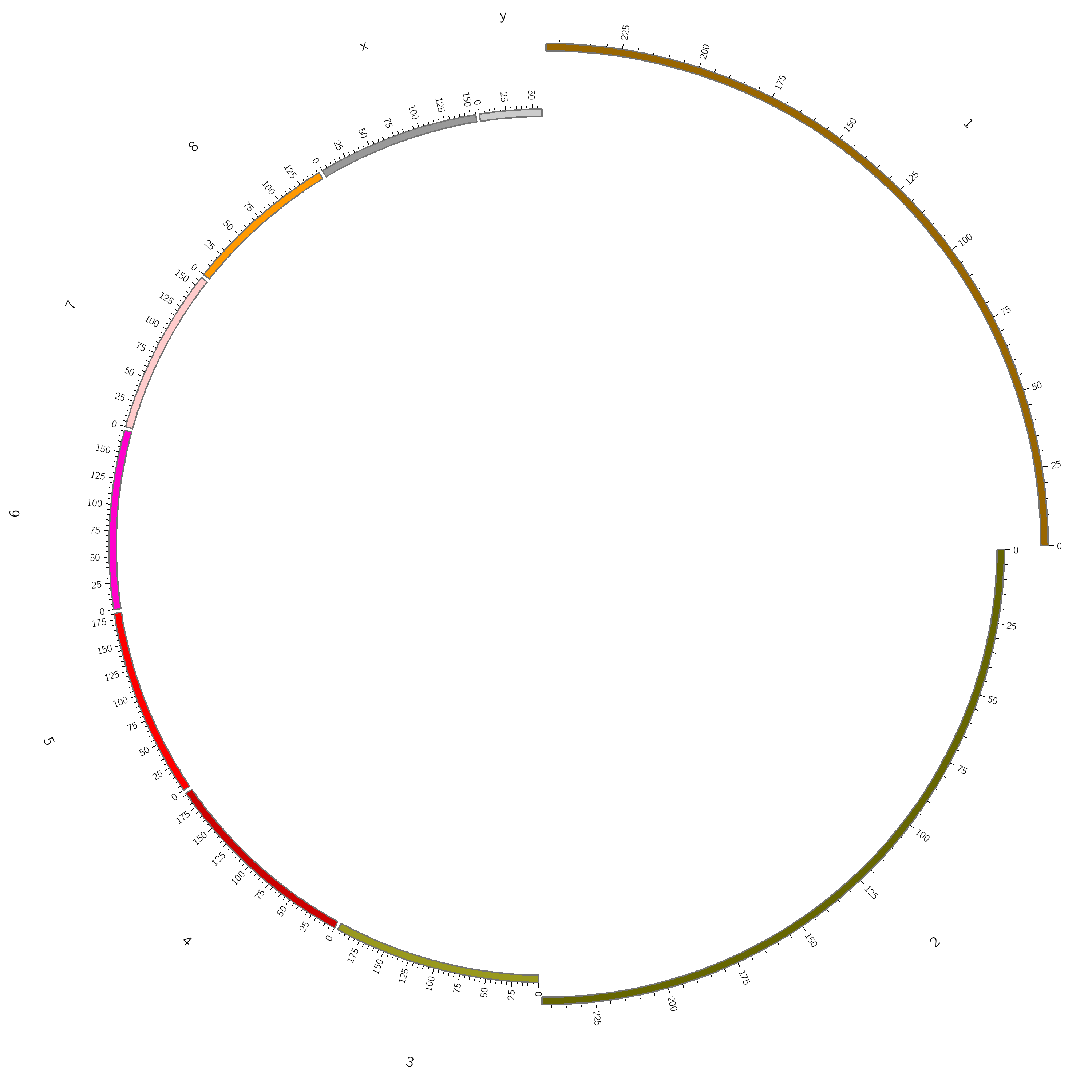
Ticks
Ticks主要是指坐标刻度和区块分隔线等,可以针对不同尺度设置不同的刻度显示方式。需要注意的是,Circos是由大至小进行刻度标识的,因此大尺度的刻度显示方式会替换小尺度。这里我们新建一个ticks.conf文件来设置相关信息
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
| # 是否显示ticks
show_ticks = yes
# 是否显示ticks的lables
show_tick_labels = yes
#子程序ticks的设置
<ticks>
# ticks的位置
radius = 1r
# ticks的颜色
color = black
# ticks的厚度
thickness = 2p
multiplier = 1e-6
# label 值的格式化方法。%d 表示结果为整数;%f 结果为浮点数; %.1f 结果为小数点后保留1位; %.2f 结果为小数点后保留2位。
format = %d
## 以下设置了 2 个 ticks,前者是小刻度,后者是大刻度。
<tick>
# 设置每个刻度代表的长度。若其单位为 u,则必须要设置 chromosomes_units 参数。比如设置 chromosomes_units = 1000000,则如下 5u 表示每个刻度代表 5M 长度的基因组序列。
spacing = 5u
# 设置 tick 的长度
size = 10p
</tick>
<tick>
spacing = 25u
size = 15p
# 以下用于设置展示 ticks' label。
show_label = yes
# 设置 ticks' label 的字体大小
label_size = 20p
# 设置 ticks' label 离 ticks 的距离
label_offset = 10p
format = %d
</tick>
</ticks>
|
此外需要将上一个板块儿中的参数50p改为250p,否则ticks会与染色体的label重叠
1
| label_radius = dims(ideogram,radius_outer) + 250p
|
最终我们就得到的圈图,如下图所示
![8RAfmQ.png]()
最后我们的配置circos.conf文件就变成了
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
| #karyotypes
<<include karyotype.conf>>
# Ideograms
<<include ideogram.conf>>
# Image
<image>
<<include etc/image.conf>>
</image>
#Ticks
<<include ticks.conf>>
# RGB/HSV color definitions, color lists, location of fonts, fill patterns.
<<include etc/colors_fonts_patterns.conf>>
# Debugging, I/O and other system parameters
<<include etc/housekeeping.conf>>
|